Unha investigación liderada polo profesor da USC Antonio Salas e Federico Martinón, xefe de servizo de Pediatría do Complexo Hospitalario de Santiago, salienta o impacto na pandemia de coronavirus de persoas con alta sensibilidade para transmitir o virus COVID-19, denominadas "supercontaxiadoras". Estas persoas poderían ter provocado entre un terzo e a metade de todos os casos rexistrados no planeta, segundo o equipo, que analizou case 5.000 xenomas do coronavirus recollidos en todo o mundo.
O equipo analizou case 5.000 xenomas do coronavirus
O equipo, do que tamén forman parte Alberto Gómez Carballa, Xabi Belo e Jacobo Pardo Seco, publicou este xoves os primeiros resultados da súa investigación. Antonio Salas explica que ata agora esta figura do ‘supercontaxiador’ discutírase nos medios e desde un punto de vista epidemiolóxico "sen outra evidencia", pero que agora esta investigación conseguiu revelar probas da súa existencia. Nalgúns lugares, estas figuras deron lugar ao que os xenetistas denominan tecnicamente como efectos fundadores locais, que se traduciron en brotes epidémicos locais ou nacionais.
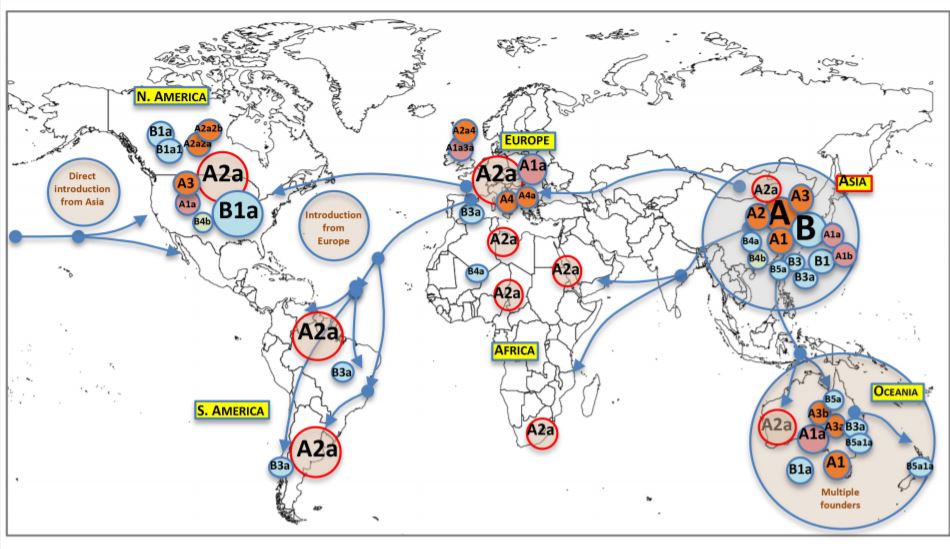
Os investigadores explican que en España entraron as primeiras cepas do virus que afectaron a case toda Europa "pero a maiores, recibimos unha cepa asiática que apenas entrou en ningún outro país europeo"
En palabras do doutor Salas, “este é un paso fundamental para entender o proceso de dispersión do virus; e seranos de grande utilidade para tratar de prever e previr futuros brotes e pandemias, xa sexa de coronavirus ou outros patóxenos con potencial igualmente letal ou mesmo superior”. Salas engade que “o escenario en España é un tanto particular no contexto do continente; no noso país entraron as primeiras cepas do virus que afectaron a case toda Europa, pero a maiores, recibimos unha cepa asiática que apenas entrou en ningún outro país europeo; un ‘supercontaxiador’ pertencente, especificamente, á liñaxe B3a”.
Antonio Salas, profesor da Facultade de Medicina da USC, afirma que “tiñamos claro que para entender o que estaba a ocorrer nesta pandemia primeiro debiamos facer unha reconstrución adecuada do proceso evolutivo que deu lugar ao virus e as súas distintas versións actuais; unha árbore filoxenética que relaciona todos os xenomas dunha maneira precisa e que é o alicerce fundamental sobre o que bascula case todo o demais”. "Tratábase de aproveitar toda a nosa experiencia no campo da evolución xenómica e levala ao terreo do SARS- CoV-2”, engade.
O traballo sitúa "de maneira precisa" a orixe evolutiva máis recente de todas as cepas actuais "non antes de novembro de 2019”
Ademais, empregando simulacións teóricas que se alimentan da variabilidade xenómica observada no coronavirus, o traballo sitúa "de maneira precisa" a orixe evolutiva máis recente de todas as cepas actuais "non antes de novembro de 2019”. De igual xeito, os investigadores confirman que a variabilidade xenética do coronavirus "correspóndese ao esperado por un proceso evolutivo natural". Neste senso, conclúen que “os datos non serían compatibles con manipulacións de laboratorio, baseadas exclusivamente en teorías ‘conspiracionistas’ sen argumento científico”.


